Способы получения баллов РЖД Бонус. Куда потратить баллы
Часто путешествуя на поездах Сапсан, ФПК и Аллегро, у пассажиров есть возможность копить бонусные баллы в рамках программы лояльности РЖД Бонус и потом тратить их на приобретение премиальных билетов на железнодорожный транспорт в любом направлении на территории Российской Федерации.
Содержание статьи
Как копить бонусные баллы?
Есть два способа, благодаря которым можно накапливать бонусы в рамках программы РЖД Бонус:
- Первый способ: премиальные баллы. Этим видом баллов пользователи могут расплачиваться за предоставленные премиальные услуги, которые не идут в расчет для получения золотого уровня. Эти бонусы можно получить при осуществлении поездок и покупок в вышеперечисленных составах. Есть несколько способов, как можно получить премиальные баллы:
- премиальные баллы начисляются на счет за приобретение товаров и использование услуг у партнеров программы
- премиальные баллы зачисляются участникам элитного клуба
- баллы зачисляются на счет за каждого вовлеченного участника по партнерской программе
- баллы получают участники спецпредложений и акций
- начисляются как приветственный подарок при достижении золотого уровня.

- Второй способ. Квалифицированные баллы получает каждый участник программы за совершенные и оплаченные поездки в железнодорожном транспорте АО ФПК, в поездах Аллегро, Сапсан и др. Премиальные баллы нужны для того, что подтвердить или достигнуть элитного уровня.
Бонусы начисляются на протяжении месяца с момента осуществления путешествия. У баллов нет срока действия при условии, что участник программы совершает поездку минимум 1 раз на протяжении 2 лет, то есть совершает поездку на железнодорожном транспорте или оплачивает билет с указанием личного счета при приобретении проездного документа. За совершение льготных поездок бонусные баллы на счет не начисляются.
Как узнать, какое число бонусных баллов находится на счету РЖД Бонус?
Проверить количество накопленных баллов можно двумя способами:
- по номеру телефона горячей линии 8 800 1001000?
- через интернет.
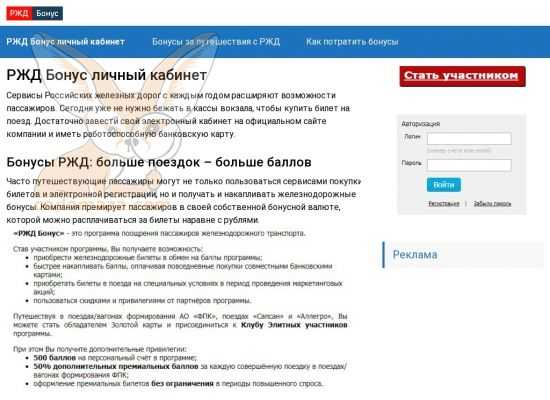
Как проверить баланс бонусных баллов через личный кабинет?
Что проверить количество бонусных баллов на своем счету через интернет, достаточно перейти на сайт rzd-bonus.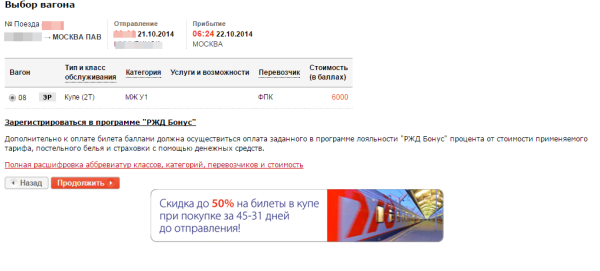 ru, в правой стороне будет меню с разделом «Авторизация», там нужно ввести пароль и логин (номер счета или почтовый адрес) для входа в личный кабинет и нажать на команду «Войти».
ru, в правой стороне будет меню с разделом «Авторизация», там нужно ввести пароль и логин (номер счета или почтовый адрес) для входа в личный кабинет и нажать на команду «Войти».
Вся информация по накопленным баллам будет отображена на главной странице личного кабинета, отдельно будут отображены квалифицированные и премиальные баллы.
Как тратить бонусные баллы в рамках программы РЖД Бонус?
Тратить накопленные баллы можно при совершении оплаты проездных документов в вагонах класса СВ, Люкс, «Купе». Проездные билеты могут оформляться на участника программы, а также на любое другое лицо. Оформлять билеты можно на сайте: pass.rzd.ru/main-pass/public/ru.
Как приобрести железнодорожный билет РЖД Бонус?
Как уже говорилось ранее, покупать билеты на железнодорожный транспорт можно на сайте pass.rzd.ru. Бонусными баллами можно рассчитываться за проездные документы на поезда «Аллегро», Сапсан, АО «ФПК».
Получить премиальный билет можно на главной странице сайта в блоке «Программы лояльности «РЖД Бонус», где нужно нажать на красную опцию «Купить премиальный билет». В появившемся окне введите следующую информацию:
В появившемся окне введите следующую информацию:
- пароль для входа в систему
- точку отправления
- ввести номер счета в программе лояльности (чтобы максимально упростить процедуру заказа билетов в будущем, нужно поставить галочку возле поля «Запомнить номер». Это нужно для того, чтобы в следующий раз не вводить пароль и логин снова и снова)
- выбрать дату поездки
- ввести место прибытия.
После введения всех данных, нажмите на опцию «Купить билет». Следите за тем, чтобы на текущем счету было необходимое число бонусов для оплаты премиального билета, который был выбран пользователем, а также минимум одно начисление бонусных баллов по факту осуществленной поездки на протяжении двух последних лет.
Преимущества пептидов RGD для направления ассоциации клеток с биоматериалами
1. Hynes RO. Интегрины: двунаправленные аллостерические сигнальные машины. Клетка. 2002; 110: 673–687. [PubMed] [Google Scholar]
2. Liddington RC, Ginsberg MH. Активация интегрина обретает форму. Джей Селл Биол. 2002; 158: 833–839. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Liddington RC, Ginsberg MH. Активация интегрина обретает форму. Джей Селл Биол. 2002; 158: 833–839. [Бесплатная статья PMC] [PubMed] [Google Scholar]
3. Arnaout MA, Mahalingam B, Xiong JP. Структура интегрина, аллостерия и двунаправленная передача сигналов. Annu Rev Cell Dev Biol. 2005; 21: 381–410. [PubMed] [Академия Google]
4. Фон дер Марк К., Парк Дж. Ю., Бауэр С., Шмуки П. Наноинженерия биомиметических поверхностей: сигналы внеклеточного матрикса. Сотовые Ткани Res. 2010; 339:131–153. [PubMed] [Google Scholar]
5. Barczyk M, Carracedo S, Gullberg D. Integrins. Сотовые Ткани Res. 2010; 339: 269–280. [Бесплатная статья PMC] [PubMed] [Google Scholar]
6. Plough EF, Haas TA, Zhang L, Loftus J, Smith JW. Связывание лиганда с интегринами. Дж. Биол. Хим. 2000; 275:21785–21788. [PubMed] [Академия Google]
7. Роуч П., Фаррар Д., Перри К.С. Интерпретация адсорбции белка: поверхностные конформационные изменения. J Am Chem Soc. 2005; 127:8168–8173. [PubMed] [Google Scholar]
8. Роуч П., Фаррар Д., Перри К.С. Адаптация поверхности для контролируемой адсорбции белка: влияние топографии в нанометровом масштабе и химия. J Am Chem Soc. 2006; 128:3939–3945. [PubMed] [Google Scholar]
Роуч П., Фаррар Д., Перри К.С. Адаптация поверхности для контролируемой адсорбции белка: влияние топографии в нанометровом масштабе и химия. J Am Chem Soc. 2006; 128:3939–3945. [PubMed] [Google Scholar]
9. Shen JW, Wu T, Wang Q, Pan HH. Молекулярное моделирование адсорбции и десорбции белков на поверхности гидроксиапатита. Биоматериалы. 2008;29: 513–532. [PubMed] [Google Scholar]
10. Courtney JM, Zhao XB, Qian H, Sharma A. Модификация полимерных поверхностей: оптимизация подходов. Перфузия. 2003; 18:33–39. [PubMed] [Google Scholar]
11. Allen LT, Tosetto M, Miller IS, O’Conner DP, Penney SC, Lynch I, et al. Поверхностно-индуцированные изменения в адсорбции белка и последствия для клеточных фенотипических ответов на взаимодействие с поверхностью. Биоматериалы. 2006; 27:3096–3108. [PubMed] [Google Scholar]
12. Кеселовский Б.Г., Коллард Д.М., Гарсия А.Дж. Химия поверхности модулирует конформацию фибронектина и направляет связывание и специфичность интегрина для контроля клеточной адгезии. J Biomed Mater Res A. 2003; 66: 247–259.. [PubMed] [Google Scholar]
J Biomed Mater Res A. 2003; 66: 247–259.. [PubMed] [Google Scholar]
13. Wilson CJ, Clegg RE, Leavesley DI, Pearcy MJ. Опосредование взаимодействий биоматериал-клетка адсорбированными белками: обзор. Ткань англ. 2005; 11:1–18. [PubMed] [Google Scholar]
14. Woods A, Couchman JR, Johansson S, Hook M. Адгезия и организация цитоскелета фибробластов в ответ на фрагменты фибронектина. EMBO J. 1986; 5: 665–670. [Бесплатная статья PMC] [PubMed] [Google Scholar]
15. Аота С., Нагаи Т., Ямада К.М. Характеристика областей фибронектина помимо последовательности аргинин-глицин-аспарагиновая кислота, необходимой для адгезивной функции клеточно-связывающего домена, с использованием сайт-направленного мутагенеза. Дж. Биол. Хим. 1991;266:15938–15943. [PubMed] [Google Scholar]
16. Хаутанен А., Гайлит Дж., Манн Д.М., Руослахти Э. Влияние модификаций последовательности RGD и ее контекста на распознавание рецептором фибронектина. Дж. Биол. Хим. 1989; 264:1437–1442. [PubMed] [Google Scholar]
17. Аота С., Номидзу М., Ямада К.М. Короткая аминокислотная последовательность Pro-His-Ser-Arg-Asn в человеческом фибронектине усиливает клеточно-адгезивную функцию. Дж. Биол. Хим. 1994; 269:24756–24761. [PubMed] [Google Scholar]
Аота С., Номидзу М., Ямада К.М. Короткая аминокислотная последовательность Pro-His-Ser-Arg-Asn в человеческом фибронектине усиливает клеточно-адгезивную функцию. Дж. Биол. Хим. 1994; 269:24756–24761. [PubMed] [Google Scholar]
18. Bowditch RD, Hariharan M, Tominna EF, Smith JW, Yamada KM, Getzoff ED, et al. Идентификация нового сайта связывания интегрина в фибронектине. Дифференциальное использование бета-3 интегринов. Дж. Биол. Хим. 1994;269:10856–10863. [PubMed] [Google Scholar]
19. Нагаи Т., Ямакава Н., Аота С., Ямада С.С., Акияма С.К., Олден К. и др. Характеристика моноклональных антител двух удаленных участков, необходимых для функции центрального клеточно-связывающего домена фибронектина в клеточной адгезии, миграции клеток и сборке матрикса. Джей Селл Биол. 1991; 114:1295–1305. [Бесплатная статья PMC] [PubMed] [Google Scholar]
20. Mao Y, Schwarzbauer JE. Доступность сайта синергии фибронектина в 3D-матрице регулирует взаимодействие интегриновых рецепторов альфа5бета1 по сравнению с альфавбета3. Клетки сотовой связи. 2006; 13: 267–277. [PubMed] [Академия Google]
Клетки сотовой связи. 2006; 13: 267–277. [PubMed] [Академия Google]
21. Руослахти Э. История РГД: личный кабинет. Матрица биол. 2003; 22: 459–465. [PubMed] [Google Scholar]
22. Plopper GE, McNamee HP, Dike LE, Bojanowski K, Ingber DE. Конвергенция сигнальных путей интегрина и рецептора фактора роста в комплексе фокальной адгезии. Мол Биол Селл. 1995; 6: 1349–1365. [Бесплатная статья PMC] [PubMed] [Google Scholar]
23. Tran KT, Griffith L, Wells A. Передача сигналов внеклеточного матрикса через рецепторы фактора роста во время заживления ран. Восстановление ран. 2004; 12: 262–268. [PubMed] [Академия Google]
24. Феррис Д.М., Муди Г.Д., Даймонд П.М., Джоранни К.В., Эрлих М.Г., Валентини Р.Ф. Титановые имплантаты с RGD-покрытием стимулируют усиление костеобразования in vivo. Биоматериалы. 1999;20:2323–2331. [PubMed] [Google Scholar]
25. Eid K, Chen E, Griffith LG, Glowacki J. Влияние RGD-покрытия на остеосовместимость PLGA-полимерных дисков в ране большеберцовой кости крысы. J Biomed Mater Res. 2001; 57: 224–231. [PubMed] [Google Scholar]
J Biomed Mater Res. 2001; 57: 224–231. [PubMed] [Google Scholar]
26. Альсберг Э., Андерсон К.В., Альбейрути А., Роули Дж.А., Муни Д.Дж. Инженерные растущие ткани. Proc Natl Acad Sci. 2002;99:12025–12030. [Бесплатная статья PMC] [PubMed] [Google Scholar]
27. Schense JC, Bloch J, Aebischer P, Hubbell JA. Ферментативное включение биоактивных пептидов в матрицу фибрина усиливает удлинение нейритов. Нац биотехнолог. 2000;18:415–419. [PubMed] [Google Scholar]
28. Yu X, Bellamkonda RV. Тканеинженерные каркасы являются эффективной альтернативой аутотрансплантатам для перекрытия промежутков периферических нервов. Ткань англ. 2003; 9: 421–430. [PubMed] [Google Scholar]
29. Li F, Carlsson D, Lohmann C, Suuronen E, Vascotto S, Kobuch K, et al. Регенерация клеток и нервов в биосинтетическом внеклеточном матриксе для трансплантации роговицы. Proc Natl Acad Sci. 2003; 100:15346–15351. [Бесплатная статья PMC] [PubMed] [Google Scholar]
30. Петри Т.А., Рейнор Дж. Э., Рейес К.Д., Бернс К.Л., Коллард Д.М., Гарсия А.Дж. Влияние интегрин-специфических биоактивных покрытий на заживление тканей и остеоинтеграцию имплантатов. Биоматериалы. 2008; 29: 2849–2857. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Э., Рейес К.Д., Бернс К.Л., Коллард Д.М., Гарсия А.Дж. Влияние интегрин-специфических биоактивных покрытий на заживление тканей и остеоинтеграцию имплантатов. Биоматериалы. 2008; 29: 2849–2857. [Бесплатная статья PMC] [PubMed] [Google Scholar]
31. Schliephake H, Scharnweber D, Dard M, Rossler S, Sewing A, Meyer J, et al. Влияние RGD-пептидного покрытия титановых имплантатов на формирование кости вокруг имплантата в альвеолярном гребне. Экспериментальное пилотное исследование на собаках. Clin Oral Implants Res. 2002; 13:312–319. [PubMed] [Google Scholar]
32. Барбер Т.А., Хо Дж.Е., Де Раньери А., Вирди А.С., Самнер Д.Р., Хили К.Е. Формирование кости вокруг имплантата и прочность интеграции имплантата с модифицированными пептидами титановыми имплантатами p(AAM-co-EG/AAC) с взаимопроникающим полимерным сетчатым покрытием. J Biomed Mater Res A. 2007; 80: 306–320. [PubMed] [Google Scholar]
33. Hennessy KM, Clem WC, Phipps MC, Sawyer AA, Shaikh FM, Bellis SL. Влияние RGD-пептидов на остеоинтеграцию гидроксиапатитных биоматериалов. Биоматериалы. 2008;29: 3075–3083. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Биоматериалы. 2008;29: 3075–3083. [Бесплатная статья PMC] [PubMed] [Google Scholar]
34. Kilpadi KL, Chang PL, Bellis SL. Гидроксиапатит связывает больше белков сыворотки, очищенных интегринов и клеток-предшественников остеобластов, чем титан или сталь. J Biomed Mater Res. 2001; 57: 258–267. [PubMed] [Google Scholar]
35. Kilpadi KL, Sawyer AA, Prince CW, Chang PL, Bellis SL. Первичные стромальные клетки костного мозга человека и клетки остеосаркомы Saos-2 используют разные механизмы для прикрепления к гидроксиапатиту. J Biomed Mater Res A. 2004; 68: 273–285. [PubMed] [Академия Google]
36. Сойер А.А., Хеннесси К.М., Беллис С.Л. Влияние адсорбированных сывороточных белков, RGD и протеогликан-связывающих пептидов на адгезию мезенхимальных стволовых клеток к гидроксиапатиту. Биоматериалы. 2007; 28: 383–392. [PubMed] [Google Scholar]
37. Сойер А.А., Хеннесси К.М., Беллис С.Л. Регуляция прикрепления и распространения мезенхимальных стволовых клеток на гидроксиапатите с помощью RGD-пептидов и адсорбированных белков сыворотки. Биоматериалы. 2005; 26:1467–1475. [PubMed] [Google Scholar]
Биоматериалы. 2005; 26:1467–1475. [PubMed] [Google Scholar]
38. Hennessy KM, Pollot BE, Clem WC, Phipps MC, Sawyer AA, Culpepper BK, et al. Влияние пептидов-миметиков коллагена I на адгезию и дифференцировку мезенхимальных стволовых клеток, а также на формирование кости на поверхности гидроксиапатита. Биоматериалы. 2009 г.;30:1898–1909. [Бесплатная статья PMC] [PubMed] [Google Scholar]
39. Lin HB, Sun W, Mosher DF, Garcia-Echeverria C, Schaufelberger K, Lelkes PI, et al. Синтез, поверхностные и клеточно-адгезионные свойства полиуретанов, содержащих ковалентно привитые RGD-пептиды. J Biomed Mater Res. 1994; 28: 329–342. [PubMed] [Google Scholar]
40. Beer JH, Springer KT, Coller BS. Иммобилизованные пептиды Arg-Gly-Asp (RGD) различной длины в качестве структурных зондов рецептора гликопротеина IIb/IIIa тромбоцитов. Кровь. 1992;79:117–128. [PubMed] [Google Scholar]
41. Lin HB, Garcia-Echeverria C, Asakura S, Sun W, Mosher DF, Cooper SL. Адгезия эндотелиальных клеток на полиуретанах, содержащих ковалентно присоединенные RGD-пептиды.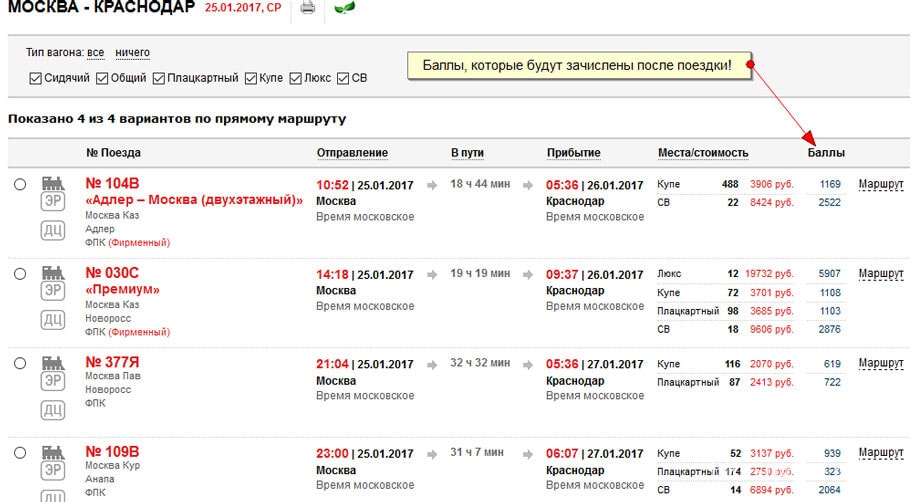 Биоматериалы. 1992; 13: 905–914. [PubMed] [Google Scholar]
Биоматериалы. 1992; 13: 905–914. [PubMed] [Google Scholar]
42. Pierschbacher M, Ruoslahti E. Влияние стереохимии последовательности Arg-Gly-Asp-Xaa на специфичность связывания при клеточной адгезии. Дж. Биол. Хим. 1987; 262:17294–17298. [PubMed] [Google Scholar]
43. Плуг Э.Ф., Пиршбахер М.Д., Руослахти Э., Маргери Г., Гинзберг М.Х. Последовательности аргинил-глицил-аспарагиновой кислоты и связывание фибриногена с тромбоцитами. Кровь. 1987;70:110–115. [PubMed] [Google Scholar]
44. Aumailley M, Gurrath M, Muller G, Calvete J, Timpl R, Kessler H. Arg-Gly-Asp, ограниченный циклическими пентапептидами. Сильные и селективные ингибиторы клеточной адгезии к витронектину и фрагменту ламинина Р1. ФЭБС лат. 1991; 291:50–54. [PubMed] [Google Scholar]
45. Хаубнер Р., Гратиас Р., Дифенбах Б., Гудман С.Л., Джончик А., Кесслер Х. Структурные и функциональные аспекты RGD-содержащих циклических пентапептидов как сильнодействующих и селективных антагонистов интегрина альфа-бета3. J Am Chem Soc. 1996;118:7461–7472. [Google Scholar]
J Am Chem Soc. 1996;118:7461–7472. [Google Scholar]
46. Tranqui L, Andrieux A, Hudry-Clergeon G, Ryckewart JJ, Soyez S, Chapel A, et al. Дифференциальные структурные требования для связывания фибриногена с тромбоцитами и эндотелиальными клетками. Джей Селл Биол. 1989; 108: 2519–2527. [Бесплатная статья PMC] [PubMed] [Google Scholar]
47. Хекманн Д., Кесслер Х. Дизайн и химический синтез интегриновых лигандов. Методы Энзимол. 2007; 426:463–503. [PubMed] [Google Scholar]
48. Hersel U, Dahmen C, Kessler H. Полимеры, модифицированные RGD: биоматериалы для стимулированной клеточной адгезии и не только. Биоматериалы. 2003; 24:4385–4415. [PubMed] [Академия Google]
49. Discher DE, Janmey P, Wang YL. Тканевые клетки чувствуют и реагируют на жесткость своего субстрата. Наука. 2005; 310:1139–1143. [PubMed] [Google Scholar]
50. Мясник Д.Т., Аллистон Т., Уивер В.М. Напряженная ситуация: форсирование опухолевой прогрессии. Нат Рев Рак. 2009; 9: 108–122. [Бесплатная статья PMC] [PubMed] [Google Scholar]
51. Данилов Ю.Н., Джулиано Р.Л. Конъюгаты (Arg-Gly-Asp)n-альбумина как модель субстрата для интегрин-опосредованной клеточной адгезии. Разрешение ячейки опыта. 1989; 182: 186–19.6. [PubMed] [Google Scholar]
Данилов Ю.Н., Джулиано Р.Л. Конъюгаты (Arg-Gly-Asp)n-альбумина как модель субстрата для интегрин-опосредованной клеточной адгезии. Разрешение ячейки опыта. 1989; 182: 186–19.6. [PubMed] [Google Scholar]
52. Arnold M, Cavalcanti-Adam EA, Glass R, Blummel J, Eck W, Kantlehner M, et al. Активация функции интегрина адгезивными интерфейсами с наноструктурой. Хемофиз. 2004; 5: 383–388. [PubMed] [Google Scholar]
53. Arnold M, VCH-W, Lohmuller T, Heil P, Blummel J, Cavalcanti-Adam EA, et al. Индукция поляризации и миграции клеток градиентом наноразмерных вариаций расстояния между адгезивными лигандами. Нано Летт. 2008; 8: 2063–2069. [Бесплатная статья PMC] [PubMed] [Google Scholar]
54. Кавальканти-Адам Э.А., Микулет А., Блюммель Дж., Ауэрнхаймер Дж., Кесслер Х., Шпатц Дж.П. Латеральное расстояние между интегриновыми лигандами влияет на распространение клеток и сборку фокальной адгезии. Eur J Cell Biol. 2006; 85: 219–224. [PubMed] [Google Scholar]
55. Cavalcanti-Adam EA, Volberg T, Micoulet A, Kessler H, Geiger B, Spatz JP. Распространение клеток и динамика фокальной адгезии регулируются расстоянием между интегриновыми лигандами. Биофиз Дж. 2007; 92: 2964–2974. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Распространение клеток и динамика фокальной адгезии регулируются расстоянием между интегриновыми лигандами. Биофиз Дж. 2007; 92: 2964–2974. [Бесплатная статья PMC] [PubMed] [Google Scholar]
56. Махешвари Г., Браун Г., Лауффенбургер Д.А., Уэллс А., Гриффит Л.Г. Клеточная адгезия и подвижность зависят от кластеризации наноразмерных RGD. Дж. Клеточные науки. 2000; 113:1677–1686. [PubMed] [Google Scholar]
57. Массия СП. Расстояние RGD в 440 нм достаточно для опосредованного интегрином альфа V бета 3 распространения фибробластов и 140 нм для фокального контакта и образования стрессовых волокон. Джей Селл Биол. 1991; 114:1089. [Бесплатная статья PMC] [PubMed] [Google Scholar]
58. Lee KY, Alsberg E, Hsiong S, Comisar W, Linderman J, Ziff R, et al. Организация лиганда наноразмерной адгезии регулирует пролиферацию и дифференцировку остеобластов. Нано Летт. 2004; 4: 1501–1506. [Бесплатная статья PMC] [PubMed] [Google Scholar]
59. Koo LY, Irvine DJ, Mayes AM, Lauffenburger DA, Griffith LG. Совместная регуляция клеточной адгезии с помощью наноразмерной организации RGD и механического стимула. Дж. Клеточные науки. 2002; 115:1423–1433. [PubMed] [Google Scholar]
Совместная регуляция клеточной адгезии с помощью наноразмерной организации RGD и механического стимула. Дж. Клеточные науки. 2002; 115:1423–1433. [PubMed] [Google Scholar]
60. Brandley BK, Schnaar RL. Ковалентное присоединение пептида последовательности Arg-Gly-Asp к дериватизируемым полиакриламидным поверхностям: поддержка адгезии фибробластов и долгосрочный рост. Анальная биохимия. 1988; 172: 270–278. [PubMed] [Google Scholar]
61. Huang J, Grater SV, Corbellini F, Rinck S, Bock E, Kemkemer R, et al. Влияние порядка и беспорядка в непаттернах RGD на клеточную адгезию. Нано Летт. 2009 г.;9:1111–1116. [Бесплатная статья PMC] [PubMed] [Google Scholar]
62. Hojo K, Susuki Y, Maeda M, Okazaki I, Nomizu M, Kamada H, et al. Аминокислоты и пептиды, Часть 39: гибрид двухвалентного поли(этиленгликоля), содержащий активный участок (RGD) и его синергический участок (PHSRN) фибронектина. Bioorg Med Chem Lett. 2001; 11:1429–1432. [PubMed] [Google Scholar]
63. Maynard HD, Okada SY, Grubbs RH. Ингибирование адгезии клеток к фибронектину олигопептидзамещенными полинорборненами. J Am Chem Soc. 2001; 123:1275–1279.. [PubMed] [Google Scholar]
Ингибирование адгезии клеток к фибронектину олигопептидзамещенными полинорборненами. J Am Chem Soc. 2001; 123:1275–1279.. [PubMed] [Google Scholar]
64. Aucoin L, Griffith CM, Pleizier G, Deslandes Y, Sheardown H. Взаимодействие эпителиальных клеток роговицы и поверхностей, модифицированных комбинациями пептидов клеточной адгезии. J Biomater Sci Polym Ed. 2002; 13: 447–462. [PubMed] [Google Scholar]
65. Ochsenhirt SE, Kokkoli E, McCarthy JB, Tirrell M. Влияние вторичной структуры RGD и сайта синергии PHSRN на клеточную адгезию, распространение и специфическое участие интегрина. Биоматериалы. 2006; 27:3863–3874. [PubMed] [Академия Google]
66. Бове Д.М., Элл Б.Дж., Маквортер А.Р., Рапрегер А.С. Синдекан-1 регулирует активацию интегринов alphavbeta3 и alphavbeta5 во время ангиогенеза и блокируется синстатином, новым пептидным ингибитором. J Эксперт Мед. 2009; 206: 691–705. [Бесплатная статья PMC] [PubMed] [Google Scholar]
67. Страхи CY, Вудс А. Роль синдеканов в заболеваниях и заживлении ран. Матрица биол. 2006; 25: 443–456. [PubMed] [Google Scholar]
Матрица биол. 2006; 25: 443–456. [PubMed] [Google Scholar]
68. Dee KC, Anderson TT, Bizios R. Дизайн и функция новых остеобласт-адгезивных пептидов для химической модификации биоматериалов. J Biomed Mater Res. 1998;40:371–377. [PubMed] [Google Scholar]
69. Деттин М., Конкони М.Т., Гамбаретто Р., Паскуато А., Фолин М., Ди Белло С. и соавт. Новые остеобласт-адгезивные пептиды для стоматологических/ортопедических биоматериалов. J Biomed Mater Res. 2002; 60: 466–471. [PubMed] [Google Scholar]
70. Sagnella S, Anderson E, Sanabria N, Marchant RE, Kottke-Marchant K. Взаимодействие эндотелиальных клеток человека с биомиметическими полимерами сурфактанта, содержащими пептидные лиганды из гепарин-связывающего домена фибронектина. Ткань англ. 2005; 11: 226–236. [Бесплатная статья PMC] [PubMed] [Google Scholar]
71. Хили К.Е., Резания А., Стайл Р.А. Разработка биоматериалов для направления биологических реакций. Энн NY Acad Sci. 1999; 875: 24–35. [PubMed] [Google Scholar]
72. Резания А., Хили К.Е. Биомиметические пептидные поверхности, которые регулируют адгезию, распространение, организацию цитоскелета и минерализацию матрикса, отложенного остеобластоподобными клетками. Биотехнологическая прог. 1999; 15:19–32. [PubMed] [Google Scholar]
Резания А., Хили К.Е. Биомиметические пептидные поверхности, которые регулируют адгезию, распространение, организацию цитоскелета и минерализацию матрикса, отложенного остеобластоподобными клетками. Биотехнологическая прог. 1999; 15:19–32. [PubMed] [Google Scholar]
73. Comisar WA, Kazmers NH, Mooney DJ, Linderman JJ. Разработка гидрогелей с наноструктурным рисунком RGD для управления поведением преостеобластов: комбинированный вычислительный и экспериментальный подход. Биоматериалы. 2007;28:4409–4417. [PubMed] [Google Scholar]
74. Berg MC, Yang SY, Hammond PT, Rubner MF. Управление взаимодействием клеток млекопитающих на многослойных полиэлектролитных поверхностях с рисунком. Ленгмюр. 2004; 20:1362–1368. [PubMed] [Google Scholar]
75. Galibert M, Sancey L, Renaudet O, Coll JL, Dumy P, Boturyn D. Применение химии клик-клик для синтеза новых поливалентных конъюгатов RGD. Орг Биомол Хим. 2010;8:5133–5138. [Статья бесплатно PMC] [PubMed] [Google Scholar]
76. Thumshirn G, Hersel U, Goodman SL, Kessler H. Мультимерные циклические RGD-пептиды как потенциальные инструменты для нацеливания на опухоли: твердофазный синтез пептидов и хемоселективное лигирование оксимов. Химия. 2003;9: 2717–2725. [PubMed] [Google Scholar]
Thumshirn G, Hersel U, Goodman SL, Kessler H. Мультимерные циклические RGD-пептиды как потенциальные инструменты для нацеливания на опухоли: твердофазный синтез пептидов и хемоселективное лигирование оксимов. Химия. 2003;9: 2717–2725. [PubMed] [Google Scholar]
77. Boturyn D, Coll JL, Garanger E, Favrot MC, Dumy P. Циклопептиды, собранные в шаблон, как мультимерная система для нацеливания на интегрины и эндоцитоза. J Am Chem Soc. 2004; 126: 5730–5739. [PubMed] [Google Scholar]
78. Montet X, Funovics M, Montet-Abou K, Weissleder R, Josephson L. Мультивалентные эффекты RGD-пептидов, полученные с помощью дисплея наночастиц. J Med Chem. 2006; 49: 6087–6093. [PubMed] [Google Scholar]
79. Wangler C, Maschauer S, Prante O, Schafer M, Schirrmacher R, Bartenstein P, et al. Мультимеризация пептидов cRGD с помощью клик-химии: синтетические стратегии, химические ограничения и влияние на биологические свойства. Химбиохим. 2010;11:2168–2181. [PubMed] [Академия Google]
80. Charo IF, Nannizzi L, Phillips DR, Hsu MA, Scarborough RM. Ингибирование связывания фибриногена с GP IIb-IIIa пептидом GP IIIa. Дж. Биол. Хим. 1991; 266:1415–1421. [PubMed] [Google Scholar]
Charo IF, Nannizzi L, Phillips DR, Hsu MA, Scarborough RM. Ингибирование связывания фибриногена с GP IIb-IIIa пептидом GP IIIa. Дж. Биол. Хим. 1991; 266:1415–1421. [PubMed] [Google Scholar]
81. Cheng S, Craig WS, Mullen D, Tschopp JF, Dixon D, Pierschbacher MD. Дизайн и синтез новых циклических RGD-содержащих пептидов в качестве сильнодействующих и селективных антагонистов интегрина.альфа.IIb.бета.3. J Med Chem. 1994; 37:1–8. [PubMed] [Google Scholar]
82. Huang G, Zhou Z, Srinivasan R, Penn MS, Kottke-Marchant K, Marchant RE, et al. Аффинные манипуляции с поверхностно-конъюгированным пептидом RGD для модулирования связывания липосом с активированными тромбоцитами. Биоматериалы. 2008;29: 1676–1685. [Бесплатная статья PMC] [PubMed] [Google Scholar]
83. Wojtowicz AM, Shekaran A, Oest ME, Dupont KM, Templeman KL, Hutmacher DW, et al. Покрытие каркасов из биоматериала коллагеномиметическим пептидом GFOGER для заживления костных дефектов. Биоматериалы. 2010;31:2574–2582. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]
84. Петри Т.А., Рейес К.Д., Бернс К.Л., Гарсия А.Дж. Простое нанесение фибронектиномиметического покрытия способствует усилению остеоинтеграции титановых имплантатов. J Cell Mol Med. 2009 г.;13:2602–2612. [Бесплатная статья PMC] [PubMed] [Google Scholar]
85. Hong M, Isailovic D, McMillan RA, Conticello VP. Структура эластин-миметического полипептида с помощью твердотельного ЯМР-анализа химического сдвига. Биополимеры. 2003; 70: 158–168. [PubMed] [Google Scholar]
86. Trabbic-Carlson K, Setton LA, Chilkoti A. Набухание и механическое поведение химически сшитых гидрогелей эластиноподобных полипептидов. Биомакромолекулы. 2003; 4: 572–580. [PubMed] [Google Scholar]
87. Urry DW, Pattanaik A, Xu J, Woods TC, McPherson DT, Parker TM. Эластичные полимеры на белковой основе для наращивания и формирования мягких тканей. J Biomater Sci Polym Ed. 1998;9:1015–1048. [PubMed] [Google Scholar]
88. Halstenberg S, Panitch A, Rizzi S, Hall H, Hubbell JA. Биологически сконструированные белок-трансплантат-поли (этиленгликоль) гидрогели: клеточный адгезив и биосинтетический материал, разлагаемый плазмином, для восстановления тканей. Биомакромолекулы. 2002; 3: 710–723. [PubMed] [Google Scholar]
Биологически сконструированные белок-трансплантат-поли (этиленгликоль) гидрогели: клеточный адгезив и биосинтетический материал, разлагаемый плазмином, для восстановления тканей. Биомакромолекулы. 2002; 3: 710–723. [PubMed] [Google Scholar]
89. Liu JC, Heilshorn SC, Tirrell DA. Сравнительный ответ клеток на белки искусственного внеклеточного матрикса, содержащие домены связывания клеток RGD и CS5. Биомакромолекулы. 2004;5:497–504. [PubMed] [Google Scholar]
90. Romano NH, Sengupta D, Chung CP, Heilshorn SC. Белковые биоматериалы: наноразмерные имитаторы внеклеточного матрикса. Биохим Биофиз Акта. 2010 18 июля; [Epub перед печатью] [Бесплатная статья PMC] [PubMed] [Google Scholar]
91. Sengupta D, Heilshorn SC. Белково-инженерные биоматериалы: высоко настраиваемые каркасы тканевой инженерии. Tissue Eng, часть B, ред. 2010; 16: 285–293. [PubMed] [Google Scholar]
Обновленный портал RGD Pathway использует повышенную эффективность курирования и предоставляет расширенную информацию о пути | Геномика человека
- Геномная база данных
- Открытый доступ
- Опубликовано:
- G Thomas Hayman 1 ,
- Pushkala Jayaraman 1 ,
- Victoria Petri 1 ,
- Marek Tutaj 1 ,
- Weisong Liu 1 ,
- Jeff De Pons 1 ,
- Мелинда Р. Дуинелл 1 и
- …
- Мэри Симояма 1
4723 Доступ
7 Цитаты
4 Альтметрика
Сведения о показателях
Abstract
RGD Pathway Portal предоставляет аннотации путей для генов крысы, человека и мыши, а также диаграммы и наборы путей, все взаимосвязанные через онтологию путей. На страницах схемы представлены схема и описание, а объекты схемы связаны с дополнительными ресурсами. Недавно разработанное веб-приложение с двойной функциональностью составляет страницу диаграммы. Кураторы вводят описание, схему, ссылки и дополнительные объекты пути. Приложение объединяет их с таблицами генов путей крыс, человека и мышей, включая генетическую информацию, инструмент анализа и справочные ссылки, а также болезни, фенотип и другие аннотации путей к генам путей. Приложение увеличивает информативность страниц диаграмм, ускоряя публикацию.
На страницах схемы представлены схема и описание, а объекты схемы связаны с дополнительными ресурсами. Недавно разработанное веб-приложение с двойной функциональностью составляет страницу диаграммы. Кураторы вводят описание, схему, ссылки и дополнительные объекты пути. Приложение объединяет их с таблицами генов путей крыс, человека и мышей, включая генетическую информацию, инструмент анализа и справочные ссылки, а также болезни, фенотип и другие аннотации путей к генам путей. Приложение увеличивает информативность страниц диаграмм, ускоряя публикацию.
Обзор
Лабораторная крыса ( Rattus norvegicus ) использовалась в качестве животной модели более 150 лет, при этом инбредные линии использовались для изучения физиологии человека и многих заболеваний человека, таких как аутоиммунные, сердечно-сосудистые, почечные и легочные заболевания. метаболические, репродуктивные и урогенитальные нарушения [1]. База данных геномов крыс (RGD; http://rgd.mcw.edu) [2] представляет собой первичный архив генетических и геномных данных крыс, содержащий более 40 000 записей об активных генах крыс, а также ортологи человека и мыши. В дополнение к генам, кодирующим белок, к ним относятся гены РНК и псевдогены. Недавний анализ сообщил о 17 733 однозначных крысиных ортологах генов человека, встречающихся во многих синтетических сегментах [3], что еще раз подтверждает использование крыс в качестве модели человеческой физиологии и болезней. В RGD также размещена информация о локусах количественных признаков крыс и человека (QTL) и линиях крыс, собранных путем ручного курирования [4] с использованием расширенного набора инструментов курирования [5] и автоматизированных конвейеров. RGD использует более дюжины различных онтологий для аннотирования информации о генах, QTL и штаммах. Это одна из немногих баз данных, в которых хранятся данные человеческого QTL.
В дополнение к генам, кодирующим белок, к ним относятся гены РНК и псевдогены. Недавний анализ сообщил о 17 733 однозначных крысиных ортологах генов человека, встречающихся во многих синтетических сегментах [3], что еще раз подтверждает использование крыс в качестве модели человеческой физиологии и болезней. В RGD также размещена информация о локусах количественных признаков крыс и человека (QTL) и линиях крыс, собранных путем ручного курирования [4] с использованием расширенного набора инструментов курирования [5] и автоматизированных конвейеров. RGD использует более дюжины различных онтологий для аннотирования информации о генах, QTL и штаммах. Это одна из немногих баз данных, в которых хранятся данные человеческого QTL.
Проект Pathway Portal [6] в RGD направлен на предоставление динамической платформы, на которой пользователи могут находить ассоциации путей для генов человека, крысы и мыши. Пользователи могут получить доступ к страницам интерактивных диаграмм путей, наборам функционально связанных путей и сетей наборов, иллюстрирующих более широкие взаимодействия, для изучения связей между ними и другими доступными ресурсами.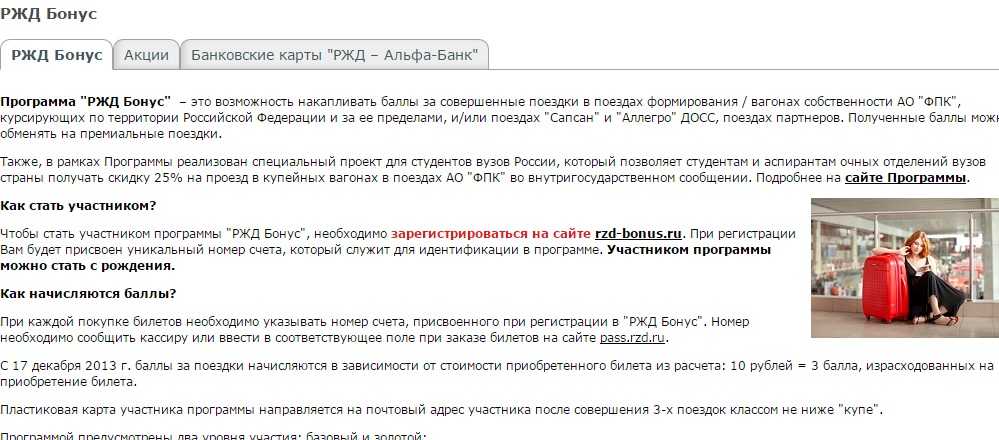 Несколько точек входа и инструментов, доступных на домашней странице RGD, позволяют пользователю искать и получать доступ к данным пути и перемещаться между компонентами портала и другими ресурсами в RGD. Поиск термина онтологии пути (PW) в поиске по общему ключевому слову RGD приведет к появлению ссылки на страницу отчета пути. Порталы заболеваний RGD также содержат ссылки на страницы отчетов о путях распространения инфекции. Курирование Pathway для Pathway Portal включает в себя аннотирование генов человека, крысы и мыши терминами в PW, которое было создано и продолжает развиваться в RGD. Кураторские данные о путях получены в основном из опубликованных научных обзоров, и нормой является аннотация к генам человека; соответствующие аннотации, сделанные для крысиных и мышиных ортологов, снабжены кодом доказательства «выведено из сходства последовательностей» (ISS), чтобы обозначить, что аннотация дополнительно предсказана для генов грызунов на основе общего сходства последовательностей с человеческим геном.
Несколько точек входа и инструментов, доступных на домашней странице RGD, позволяют пользователю искать и получать доступ к данным пути и перемещаться между компонентами портала и другими ресурсами в RGD. Поиск термина онтологии пути (PW) в поиске по общему ключевому слову RGD приведет к появлению ссылки на страницу отчета пути. Порталы заболеваний RGD также содержат ссылки на страницы отчетов о путях распространения инфекции. Курирование Pathway для Pathway Portal включает в себя аннотирование генов человека, крысы и мыши терминами в PW, которое было создано и продолжает развиваться в RGD. Кураторские данные о путях получены в основном из опубликованных научных обзоров, и нормой является аннотация к генам человека; соответствующие аннотации, сделанные для крысиных и мышиных ортологов, снабжены кодом доказательства «выведено из сходства последовательностей» (ISS), чтобы обозначить, что аннотация дополнительно предсказана для генов грызунов на основе общего сходства последовательностей с человеческим геном. Можно предположить, что использование терминов путей в разделе биологических процессов Генной онтологии (GO) будет адекватным для аннотаций путей. Хотя между PW и GO есть перекрывающиеся термины, последний использует перспективу однонаправленного процесса, чьи реакции и взаимодействия приводят к конечному результату, в то время как точка зрения PW представляет собой один из наборов взаимодействующих молекул, чьи реакции и взаимодействия лежат в основе функционирующих сетей. Эта онтология пути имеет пять основных узлов: метаболический, регуляторный, сигнальный, лекарственный и болезненный пути, с терминами для измененных версий путей. Пути введения лекарств предлагаются PharmGKB (http://www.pharmgkb.org/) и базой данных путей малых молекул (http://www.smpdb.ca/), причем последняя также имеет заболевание наряду с сигнальными и метаболическими путями. Пути развития болезни можно найти в Киотской энциклопедии генов и геномов (KEGG; http://www.genome.jp/kegg/) и Reactome (http://www.reactome.
Можно предположить, что использование терминов путей в разделе биологических процессов Генной онтологии (GO) будет адекватным для аннотаций путей. Хотя между PW и GO есть перекрывающиеся термины, последний использует перспективу однонаправленного процесса, чьи реакции и взаимодействия приводят к конечному результату, в то время как точка зрения PW представляет собой один из наборов взаимодействующих молекул, чьи реакции и взаимодействия лежат в основе функционирующих сетей. Эта онтология пути имеет пять основных узлов: метаболический, регуляторный, сигнальный, лекарственный и болезненный пути, с терминами для измененных версий путей. Пути введения лекарств предлагаются PharmGKB (http://www.pharmgkb.org/) и базой данных путей малых молекул (http://www.smpdb.ca/), причем последняя также имеет заболевание наряду с сигнальными и метаболическими путями. Пути развития болезни можно найти в Киотской энциклопедии генов и геномов (KEGG; http://www.genome.jp/kegg/) и Reactome (http://www.reactome. org/ReactomeGWT/entrypoint.html). PW RGD уникален тем, что имеет все типы путей, включая болезни и измененные (уникальные для PW) версии. Онтология позволяет стандартизировать аннотацию генов к терминам пути, обеспечивает связь с интерактивными диаграммами пути и служит инструментом навигации между различными типами данных пути. Генерация диаграмм и интерактивных страниц диаграмм являются важными компонентами процесса курирования Pathway Portal. Визуализация путей помогает пользователю понять положение и взаимосвязь между функциями генов в сетях. Приложение было разработано как для увеличения диапазона и доступности биологической информации, предоставляемой страницами схемы путей, так и для ускорения их производства.
org/ReactomeGWT/entrypoint.html). PW RGD уникален тем, что имеет все типы путей, включая болезни и измененные (уникальные для PW) версии. Онтология позволяет стандартизировать аннотацию генов к терминам пути, обеспечивает связь с интерактивными диаграммами пути и служит инструментом навигации между различными типами данных пути. Генерация диаграмм и интерактивных страниц диаграмм являются важными компонентами процесса курирования Pathway Portal. Визуализация путей помогает пользователю понять положение и взаимосвязь между функциями генов в сетях. Приложение было разработано как для увеличения диапазона и доступности биологической информации, предоставляемой страницами схемы путей, так и для ускорения их производства.
Веб-приложение страниц диаграммы молекулярного пути
Для портала пути было разработано новое приложение молекулярного пути, которое имеет две функции. Во-первых, это значительно расширяет объем информации о пути, доступной пользователю. Страницы диаграмм, сгенерированные с помощью приложения, копируются в общедоступную базу данных, где пользователи могут получить к ним доступ из точек входа Pathway на домашней странице RGD. Термины пути можно искать с помощью браузера онтологий, инструмента просмотра генома, поиска по ключевым словам или записей на порталах болезней, которые можно найти на домашней странице RGD. Результатом стал недавно пересмотренный отчет об онтологии (рис. 1) с определением и ссылкой на улучшенный браузер онтологий (рис. 1А) [7]. Полногеномная карта Genome Viewer показывает хромосомные положения генов, аннотированных на пути (рис. 1B). Список этих генов-членов пути с расположением хромосом можно загрузить через ссылку для экспорта переменных с разделителями-запятыми в нижней части средства просмотра генома с помощью программного обеспечения для работы с электронными таблицами. База данных RGD анализируется для создания таблицы, в которой есть вкладка для выбора списков генов крысы, человека или мыши, аннотированных к термину, и, если выбрано, дочерних терминов. Включена дополнительная генетическая и аннотационная информация, а также ссылка на инструмент RGD GBrowse, который теперь имеет версии для человека, крысы и мыши для дальнейшего анализа последовательности (рис.
Термины пути можно искать с помощью браузера онтологий, инструмента просмотра генома, поиска по ключевым словам или записей на порталах болезней, которые можно найти на домашней странице RGD. Результатом стал недавно пересмотренный отчет об онтологии (рис. 1) с определением и ссылкой на улучшенный браузер онтологий (рис. 1А) [7]. Полногеномная карта Genome Viewer показывает хромосомные положения генов, аннотированных на пути (рис. 1B). Список этих генов-членов пути с расположением хромосом можно загрузить через ссылку для экспорта переменных с разделителями-запятыми в нижней части средства просмотра генома с помощью программного обеспечения для работы с электронными таблицами. База данных RGD анализируется для создания таблицы, в которой есть вкладка для выбора списков генов крысы, человека или мыши, аннотированных к термину, и, если выбрано, дочерних терминов. Включена дополнительная генетическая и аннотационная информация, а также ссылка на инструмент RGD GBrowse, который теперь имеет версии для человека, крысы и мыши для дальнейшего анализа последовательности (рис.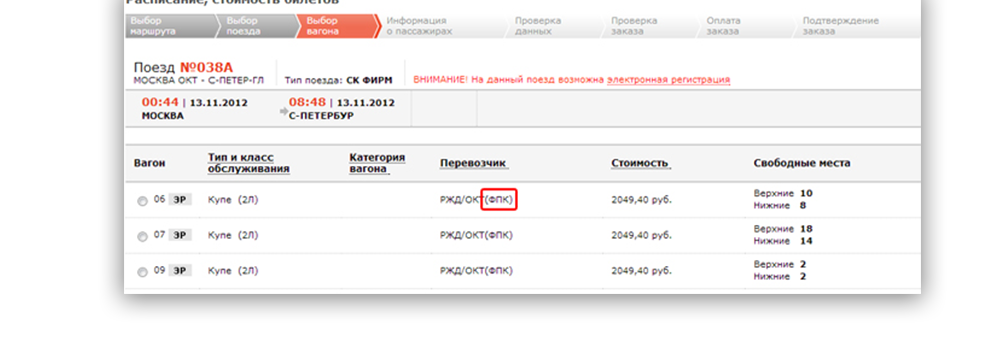 1C). Читатель отсылается к Laulederkind et al. [8] для подробного описания использования GBrowse и других инструментов анализа RGD. Таблица сортируется по нескольким параметрам. Представлен выбор представлений путей дерева онтологий для термина, включая количество существующих аннотаций на каждом уровне (рис. 1D). Если доступно, отображается значок интерактивной схемы пути (рис. 1А), который ссылается на недавно улучшенную страницу диаграммы (рис. 2). Страница схемы пути содержит расширяемое описание (рис. 2A), которое включает установленные куратором ссылки на записи Pfam для доменов, упомянутых в описании пути, таких как домены связывания фосфотирозина и домены гомологии Src, упомянутые в описании страницы отчета о сигнальном пути инсулина (не показано). Совсем недавно были предоставлены ссылки на записи банка данных о структурах Research Collaborative for Structural Bioinformatics Protein Data Bank, как на странице отчета о пути визуальной фототрансдукции, а также ссылки на записи терминов KEGG, Reactome и GO, если применимо.
1C). Читатель отсылается к Laulederkind et al. [8] для подробного описания использования GBrowse и других инструментов анализа RGD. Таблица сортируется по нескольким параметрам. Представлен выбор представлений путей дерева онтологий для термина, включая количество существующих аннотаций на каждом уровне (рис. 1D). Если доступно, отображается значок интерактивной схемы пути (рис. 1А), который ссылается на недавно улучшенную страницу диаграммы (рис. 2). Страница схемы пути содержит расширяемое описание (рис. 2A), которое включает установленные куратором ссылки на записи Pfam для доменов, упомянутых в описании пути, таких как домены связывания фосфотирозина и домены гомологии Src, упомянутые в описании страницы отчета о сигнальном пути инсулина (не показано). Совсем недавно были предоставлены ссылки на записи банка данных о структурах Research Collaborative for Structural Bioinformatics Protein Data Bank, как на странице отчета о пути визуальной фототрансдукции, а также ссылки на записи терминов KEGG, Reactome и GO, если применимо. Сама диаграмма содержит ссылки на страницы отчетов о генах, полученные из значков генов, изображенных в пути (рис. 2B), а также из той же таблицы генов-членов Genes in Pathway, представленной в отчете об онтологии (рис. 1C и рисунок 2C). Дополнительные новые списки других элементов пути, таких как члены семейства генов или гены-мишени (отдельные записи ссылаются на страницы отчета о генах), а также описания и ссылки PubChem или химических объектов биологического интереса (ChEBI) для малых молекул (рис. 2D; значки молекул на диаграмме также ссылаются на эту информацию). Таблицы, которые автоматически составляются веб-приложением, содержат проанализированную информацию о заболеваниях, других путях и фенотипах, аннотированных членам генов путей. Их можно переключать между перечислением в алфавитном порядке по гену или по заболеванию, пути или фенотипу (рис. 2E). Если пользователя интересуют один или несколько генов, эти таблицы предлагают немедленную информацию об их заболевании, фенотипе и других ассоциациях путей без необходимости переходить на страницы отдельных отчетов.
Сама диаграмма содержит ссылки на страницы отчетов о генах, полученные из значков генов, изображенных в пути (рис. 2B), а также из той же таблицы генов-членов Genes in Pathway, представленной в отчете об онтологии (рис. 1C и рисунок 2C). Дополнительные новые списки других элементов пути, таких как члены семейства генов или гены-мишени (отдельные записи ссылаются на страницы отчета о генах), а также описания и ссылки PubChem или химических объектов биологического интереса (ChEBI) для малых молекул (рис. 2D; значки молекул на диаграмме также ссылаются на эту информацию). Таблицы, которые автоматически составляются веб-приложением, содержат проанализированную информацию о заболеваниях, других путях и фенотипах, аннотированных членам генов путей. Их можно переключать между перечислением в алфавитном порядке по гену или по заболеванию, пути или фенотипу (рис. 2E). Если пользователя интересуют один или несколько генов, эти таблицы предлагают немедленную информацию об их заболевании, фенотипе и других ассоциациях путей без необходимости переходить на страницы отдельных отчетов.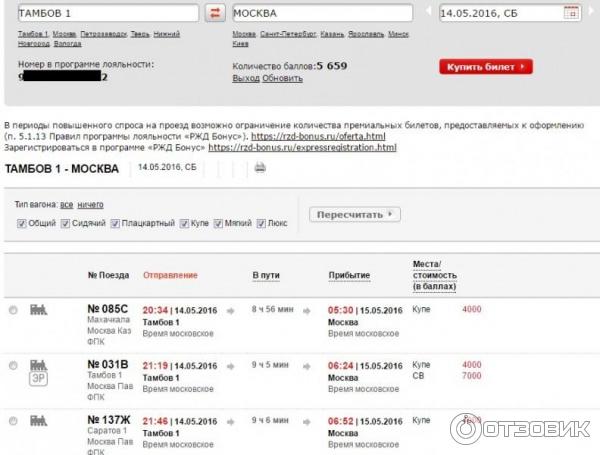 Эта информация доступна для всех генов в пути. Также предоставляется список ссылок, аннотированных термину пути (рис. 2F), и диаграмма пути онтологии, а также ссылка для загрузки диаграммы для пользователей программного обеспечения Ariadne (рис. 2G). Версию диаграммы можно сохранить, щелкнув правой кнопкой мыши фон диаграммы, а затем сохранив изображение в виде переносимого сетевого графического файла. Возможность загрузки этих списков и ассоциаций генов, а также диаграммы с помощью бесплатного программного обеспечения находится в разработке. Важно отметить, что программное обеспечение Ariadne включает расширяемую базу данных млекопитающих ResNet, которая содержит записи о генах человека, крысы и мыши, малых молекулах, заболеваниях и процессах с дополнительными данными. Тот факт, что новые функции могут быть добавлены в базу данных, был использован для создания информационных связей с малыми молекулами, описанными выше, и связей с путями, запускаемыми или связанными с исследуемым путем (рис.
Эта информация доступна для всех генов в пути. Также предоставляется список ссылок, аннотированных термину пути (рис. 2F), и диаграмма пути онтологии, а также ссылка для загрузки диаграммы для пользователей программного обеспечения Ariadne (рис. 2G). Версию диаграммы можно сохранить, щелкнув правой кнопкой мыши фон диаграммы, а затем сохранив изображение в виде переносимого сетевого графического файла. Возможность загрузки этих списков и ассоциаций генов, а также диаграммы с помощью бесплатного программного обеспечения находится в разработке. Важно отметить, что программное обеспечение Ariadne включает расширяемую базу данных млекопитающих ResNet, которая содержит записи о генах человека, крысы и мыши, малых молекулах, заболеваниях и процессах с дополнительными данными. Тот факт, что новые функции могут быть добавлены в базу данных, был использован для создания информационных связей с малыми молекулами, описанными выше, и связей с путями, запускаемыми или связанными с исследуемым путем (рис.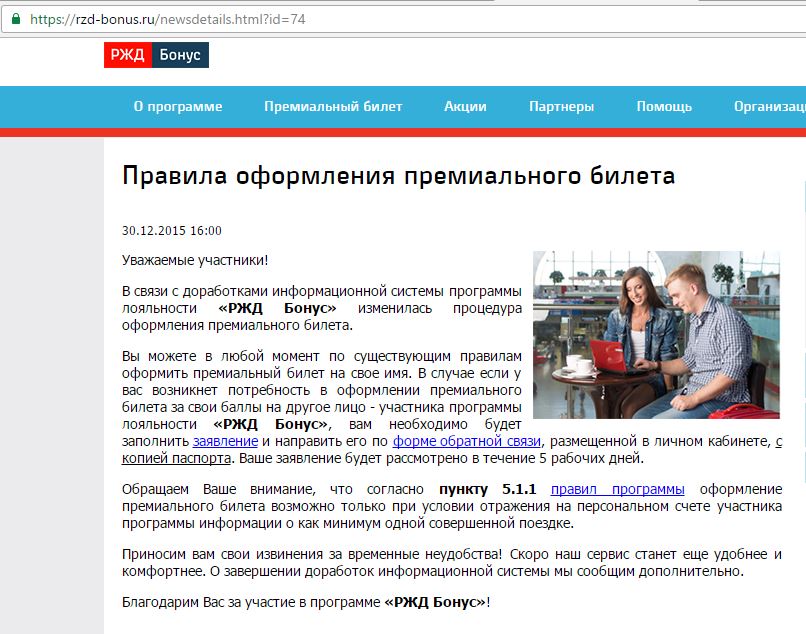 2B, путь биосинтеза тестостерона), или между путями заболевания. и основные измененные пути (рис. 2C, D, измененный сигнальный путь андрогенов). Эта функция позволяет пользователям «путешествовать» по ландшафту путей, сравнивая нормальные пути с измененными, чтобы изучить потенциальную роль, которую изменения могут играть в заболевании.
2B, путь биосинтеза тестостерона), или между путями заболевания. и основные измененные пути (рис. 2C, D, измененный сигнальный путь андрогенов). Эта функция позволяет пользователям «путешествовать» по ландшафту путей, сравнивая нормальные пути с измененными, чтобы изучить потенциальную роль, которую изменения могут играть в заболевании.
Обновлен отчет об онтологии. ( A ) Определение термина, ссылка браузера и значок диаграммы. ( B ) Дисплей просмотра генома. ( C ) Таблица членов пути гена. ( D ) Выбор пути термина онтологии с подсчетом аннотаций.
Полноразмерное изображение
Рисунок 2 Обновлена страница пути. ( A ) Расширяемое описание. ( B ) Схема пути. ( C ) Таблица членов пути гена. ( D ) Таблица дополнительных элементов пути. ( E ) Таблицы терминов болезней, других путей и фенотипов, аннотированных к генам-членам пути.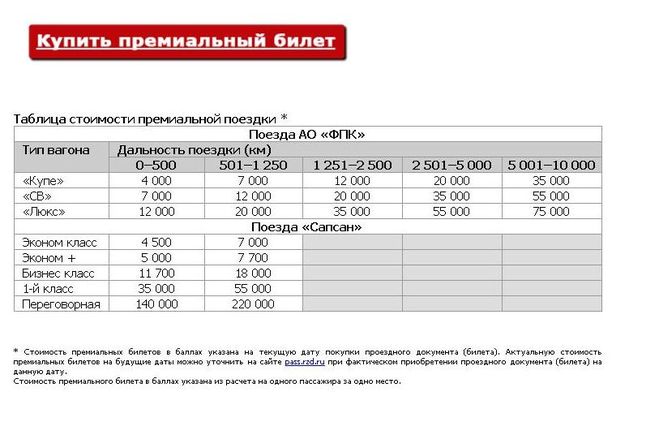 ( F ) Ссылки, аннотированные к пути. ( G ) Схема пути онтологии.
( F ) Ссылки, аннотированные к пути. ( G ) Схема пути онтологии.
Изображение в натуральную величину
Вторая функция приложения пути — значительно упростить установление кураторами множества отношений, отображаемых на страницах пути. Раньше это был сложный, детальный, запутанный и более медленный процесс. Процесс оптимизации с помощью нового приложения состоит из двух компонентов. Первый компонент — это шаблон для создания страницы отчета пути. Сюда входят описание, список связанных ссылок, связанные объекты пути и измененные пути, все из которых доступны для редактирования. Шаблон также загружает диаграмму пути. Страница ввода пути создания/редактирования (рис. 3) позволяет кураторам искать пути либо с помощью инвентарных номеров терминов PW, либо путем ввода названия термина (рис. 3А). Функция автозаполнения облегчает этот процесс. Также перечислены все пути, которые уже были созданы и сохранены в базе данных для простоты доступа (рис. 3C). Поиск пути, отсутствующего в базе данных, автоматически перенаправит страницу на форму интерфейса (см. рис. 4), что позволит куратору создать страницу диаграммы пути. Страница входа содержит предварительно сохраненную информацию об условиях пути и присоединении, извлеченную из базы данных. Поиск существующего пути с помощью первого текстового поля (рис. 3А) открывает интерфейс редактирования для этого пути. Поиск с использованием второго текстового поля (рис. 3B) приводит к отображению страницы пути (см. рис. 2). Интерфейс создания/редактирования состоит из пяти разделов (рис. 4). Описание вводится или редактируется в первом текстовом окне, а ссылки на другие базы данных внутри текста устанавливаются куратором (рис. 4А). Во втором разделе добавляются ссылки, используемые для аннотаций пути, либо в виде идентификаторов PubMed, которые автоматически преобразуются в идентификаторы RGD, либо напрямую в виде идентификаторов RGD (рис. 4B). Идентификаторы терминов для любых измененных путей вводятся в третьем разделе (рис. 4C). Дополнительные идентификаторы элементов пути, описания и ссылки PubChem или ChEBI представлены в четвертом разделе (рис.
рис. 4), что позволит куратору создать страницу диаграммы пути. Страница входа содержит предварительно сохраненную информацию об условиях пути и присоединении, извлеченную из базы данных. Поиск существующего пути с помощью первого текстового поля (рис. 3А) открывает интерфейс редактирования для этого пути. Поиск с использованием второго текстового поля (рис. 3B) приводит к отображению страницы пути (см. рис. 2). Интерфейс создания/редактирования состоит из пяти разделов (рис. 4). Описание вводится или редактируется в первом текстовом окне, а ссылки на другие базы данных внутри текста устанавливаются куратором (рис. 4А). Во втором разделе добавляются ссылки, используемые для аннотаций пути, либо в виде идентификаторов PubMed, которые автоматически преобразуются в идентификаторы RGD, либо напрямую в виде идентификаторов RGD (рис. 4B). Идентификаторы терминов для любых измененных путей вводятся в третьем разделе (рис. 4C). Дополнительные идентификаторы элементов пути, описания и ссылки PubChem или ChEBI представлены в четвертом разделе (рис. 4D). В разделах ссылок, измененных путей и дополнительных элементов пути можно создавать новые элементы или удалять существующие. Наконец, необходимые файлы и папки интерактивных диаграмм, созданные с помощью программного обеспечения Ariadne Pathway Studio (версия 8.0), загружаются для отображения (рис. 4E). Связи, установленные на диаграмме, когда она была создана с помощью программного обеспечения Ariadne, сохраняются приложением пути. Когда информация обновляется в нижней части интерфейса, куратор переходит на страницу отчета о пути (см. рис. 2), чтобы убедиться, что вся введенная информация отображается. Во втором компоненте процесса оптимизации приложение автоматически анализирует базу данных для создания информационных таблиц, описанных выше, которые отображают болезни, фенотип и другие аннотации путей, сделанные для генов в изучаемом пути. Также добавлено представление пути онтологии для термина PW.
4D). В разделах ссылок, измененных путей и дополнительных элементов пути можно создавать новые элементы или удалять существующие. Наконец, необходимые файлы и папки интерактивных диаграмм, созданные с помощью программного обеспечения Ariadne Pathway Studio (версия 8.0), загружаются для отображения (рис. 4E). Связи, установленные на диаграмме, когда она была создана с помощью программного обеспечения Ariadne, сохраняются приложением пути. Когда информация обновляется в нижней части интерфейса, куратор переходит на страницу отчета о пути (см. рис. 2), чтобы убедиться, что вся введенная информация отображается. Во втором компоненте процесса оптимизации приложение автоматически анализирует базу данных для создания информационных таблиц, описанных выше, которые отображают болезни, фенотип и другие аннотации путей, сделанные для генов в изучаемом пути. Также добавлено представление пути онтологии для термина PW.
Страница входа создания/редактирования приложения Pathway. ( A ) Идентификатор пути вводится для создания новой страницы или редактирования существующей. ( B ) Существующие страницы можно просмотреть, введя идентификатор пути. ( C ) Список существующих страниц отчета пути.
( A ) Идентификатор пути вводится для создания новой страницы или редактирования существующей. ( B ) Существующие страницы можно просмотреть, введя идентификатор пути. ( C ) Список существующих страниц отчета пути.
Полноразмерное изображение
Рисунок 4Интерфейс создания/редактирования. ( A ) Ввод/редактирование описания. ( B ) Ввод и преобразование ссылки. ( C ) Измененная запись пути. ( D ) Запись связанных объектов пути. ( E ) Загрузка схемы.
Полноразмерное изображение
Разработка программного обеспечения
Приложение Molecular Pathway построено на технологиях J2EE (http://java.sun.com/j2ee/overview.html) и управляется базой данных RGD Oracle. Его можно запустить в любом контейнере Java, который реализует спецификацию Servlet 2.4 и JavaServer Pages 2.0 или выше. Веб-приложение построено на Spring [9] Архитектура контроллера модель-представление фреймворка. Чтобы сделать ввод онтологических терминов более эффективным, была реализована функция автозаполнения онтологических терминов на основе Apache Solr (http://lucene.apache.org/solr/) и JQuery (http://jquery.com/). Эта функция также может преобразовывать термины онтологий в идентификаторы онтологий. Приложение использует асинхронный Javascript и XML (AJAX) [10], что позволяет куратору добавлять и удалять поля без обновления страницы. Кроме того, было включено быстрое извлечение AJAX, позволяющее быстро преобразовать ссылочный идентификатор PubMed в идентификатор RGD без необходимости создания новой страницы. Программа общего интерфейса шлюза (CGI), которая обрабатывает абстрактную загрузку и назначение идентификатора RGD, была изменена, чтобы программа CGI могла принимать идентификатор PubMed из интерфейса редактирования пути и автоматически отправлять результат обратно в тот же интерфейс. Эта программа не только возвращает идентификатор RGD для уже существующей ссылки, но также загружает реферат ссылки, назначает новый идентификатор RGD и возвращает вновь созданный идентификатор RGD обратно в интерфейс в случае ссылки, отсутствующей в базе данных.
Чтобы сделать ввод онтологических терминов более эффективным, была реализована функция автозаполнения онтологических терминов на основе Apache Solr (http://lucene.apache.org/solr/) и JQuery (http://jquery.com/). Эта функция также может преобразовывать термины онтологий в идентификаторы онтологий. Приложение использует асинхронный Javascript и XML (AJAX) [10], что позволяет куратору добавлять и удалять поля без обновления страницы. Кроме того, было включено быстрое извлечение AJAX, позволяющее быстро преобразовать ссылочный идентификатор PubMed в идентификатор RGD без необходимости создания новой страницы. Программа общего интерфейса шлюза (CGI), которая обрабатывает абстрактную загрузку и назначение идентификатора RGD, была изменена, чтобы программа CGI могла принимать идентификатор PubMed из интерфейса редактирования пути и автоматически отправлять результат обратно в тот же интерфейс. Эта программа не только возвращает идентификатор RGD для уже существующей ссылки, но также загружает реферат ссылки, назначает новый идентификатор RGD и возвращает вновь созданный идентификатор RGD обратно в интерфейс в случае ссылки, отсутствующей в базе данных.
Файлы Pathway Image, созданные в программном инструменте Ariadne Pathway Studio (версия 8.0), загружаются с помощью Jupload, апплета OpenSource, распространяемого на SourceForge, который позволяет загружать несколько файлов в один каталог. Этот апплет распространяется под Common Public License версии 1.0: JUnit, GNU GPL (общая общественная лицензия) V3: проект JUpload на SourceForge и Apache Software License версии 2.0: Commons Lang, Jakarta Commons Net, интерфейс прикладного программирования Maven Plugin (http ://jupload.sourceforge.net/). Скомпилированный апплет JUpload, подписанный сертификатом проекта JUpload, используется для загрузки каталогов путей на курирующий сервер. Класс Java, наследующий методы Parser и Node Visitor языка гипертекстовой разметки (HTML), используется для замены ссылок, созданных Pathway Studio, ссылками на все ресурсы RGD. Автоматическая регенерация файловой структуры гарантирует отсутствие ручного вмешательства при загрузке каждого нового или обновленного пути.
Новые объекты и обновления существующих объектов проходят через уровень проверки, чтобы уменьшить вероятность попадания ошибок в базу данных. Загруженные файлы автоматически сканируются, чтобы обеспечить загрузку правильных файлов пути. Поддерживаемые браузеры включают IE 8+, Firefox 3+ и Safari 5+. Кроме того, на страницу включены встроенные процедуры проверки, помогающие кураторам в контроле качества. Пользовательский интерфейс построен на стандартных веб-технологиях, включая HTML, JavaScript и каскадные таблицы стилей. После создания или редактирования пути куратору предоставляется «почти окончательный» вид созданного пути, который можно редактировать дальше.
Архитектура и возможности программного обеспечения Pathway Curation могут представлять интерес для сообщества информатиков. Однако, поскольку прикладное программное обеспечение пути было разработано специально для соответствия схеме базы данных RGD и использует лицензированное программное обеспечение Ariadne, его нельзя было легко адаптировать для использования с другими базами данных, поэтому оно не было общедоступным.
Перспективы и перспективы
Портал RGD Pathway продолжает расти, как и число исследователей, использующих его. Недавно был добавлен конвейер данных, импортирующий почти 24 000 аннотаций генов путей, в том числе более 8 000 для генов человека, из базы данных взаимодействия путей (http://pid.nci.nih.gov/). Аналогичный конвейер данных о пути импорта из KEGG будет выпущен в ближайшее время. В настоящее время существует более 9400 аннотаций путей и более 100 интерактивных страниц путей, опубликованных на RGD. Они представляют большой интерес для пользователей исследовательского сообщества, о чем свидетельствует количество просмотров страниц (всего 43 333, 27 547 уникальных просмотров страниц с 01 августа 2011 г. по 01 августа 2012 г.), определенное с помощью Google Analytics, а также обновления и новые тенденции, обнаруженные в других источниках данных. К ним относятся новые вебинары, посвященные инструментам визуализации и анализа пути и сети в Reactome, а также новые модули реакции в KEGG.
В процессе обеспечения динамической платформы или «ландшафта» для исследования, которым стремится быть портал Pathway, разрабатываются новые подходы или взгляды, такие как наборы путей и сети наборов. Каждый набор предлагает мгновенный снимок более широкой картины, которая объединяет несколько путей. Сети наборов соединяют связанные наборы путей, иллюстрируя их сложные взаимодействия более высокого порядка. Мы надеемся на дальнейший рост во всех этих областях. По мере того, как портал расширяется в глубину и масштабы, будут разрабатываться и добавляться другие подходы и инструменты для расширения коллекции страниц путей в RGD и ценности, которую она предлагает исследовательскому сообществу.
Сокращения
- AJAX:
асинхронный Javascript и XML
- Графика:
общий интерфейс шлюза
- ЧЭБИ:
Химические объекты, представляющие биологический интерес
- ГО:
Генная онтология
- HTML:
язык гипертекстовой разметки
- БОЧКА:
Киотская энциклопедия генов и геномов
- Пароль:
онтология пути
- РГД:
База данных генома крысы.

Ссылки
Джейкоб Х.Дж.: Функциональная геномика и модели крыс. Геном Res. 1999, 9: 1013-1016. 10.1101/гр.9.11.1013.
Артикул КАС пабмед Google ученый
Симояма М., Смит Дж. Р., Хейман Т., Лауледеркинд С., Лоури Т., Нигам Р., Петри В., Ван С.Дж., Двинелл М., Джейкоб Х., команда RGD: RGD: платформа сравнительной геномики. Гум Геномика. 2011, 5: 124-129.
Артикул ПабМед Центральный КАС пабмед Google ученый
Кемкемер С., Кон М., Купер Д.Н., Фроенике Л., Хёгель Дж., Хамейстер Х., Керер-Савацки Х.: Сравнение синтении генов между разными позвоночными дает новое понимание событий разрыва и слияния в ходе эволюции кариотипа млекопитающих. БМС Эвол Биол. 2009, 9: 84-10.1186/1471-2148-9-84.

Артикул ПабМед Центральный пабмед Google ученый
Симояма М., Хейман Г.Т., Лауледеркинд С.Дж., Нигам Р., Лоури Т.Ф., Петри В., Смит Дж.Р., Ван С.Дж., Мунценмайер Д.Х., Двинелл М.Р., Твиггер С.Н., Джейкоб Х.Дж., Team RGD: Кураторы базы данных генома крыс: кто , что, где, почему. PLoS Comput Biol. 2009, 5: e1000582-10.1371/journal.pcbi.1000582.
Артикул ПабМед Центральный пабмед Google ученый
Лауледеркинд С.Дж.Ф., Симояма М., Хейман Г.Т., Лоури Т.Ф., Нигам Р., Петри В., Смит-младший, Ван С.Дж., Де Понс Дж., Ковальски Г., Лю В., Руд В., Мунценмайер Д.Х., Дуинелл М.Р., Твиггер С.Н., Джейкоб Х.Дж., команда RGD: Набор инструментов для курирования базы данных генома крысы: набор оптимизированных программных инструментов, обеспечивающих эффективный сбор, организацию и представление биологических данных. База данных.

Артикул ПабМед Центральный пабмед Google ученый
Петри В., Симояма М., Хейман Г.Т., Смит-Дж.Р., Тутай М., Де Понс Дж., Двинелл М.Р., Мунзенмайер Д.Х., Твиггер С.Н., Джейкоб Х.Дж., Team RGD: Портал путей базы данных генома крысы. База данных. 2011, 2011: bar010-10.1093/база данных/bar010.
Артикул ПабМед Центральный пабмед Google ученый
Лауледеркинд С.Дж., Тутадж М., Шимояма М., Хейман Г.Т., Лоури Т.Ф., Нигам Р., Петри В., Смит Дж.Р., Ван С.Дж., Де Понс Дж., Двинелл М.Р., Джейкоб Х.Дж.: поиск и просмотр онтологии в базе данных генома крысы. База данных. 2012, 2012: bas016-10.1093/база данных/bas016.
Артикул ПабМед Центральный пабмед Google ученый
Laulederkind SJF, Hayman GT, Wang SJ, Lowry TF, Nigam R, Petri V, Smith JR, Shimoyama M, Dwinell MR, Jacob HJ: Изучение генетических, геномных и фенотипических данных в базе данных генома крысы.
 Протоколы Curr Bioinf. в прессе
Протоколы Curr Bioinf. в прессеУоллс C, Брайденбах R: Весна в действии. 2007, Гринвич: Публикации Мэннинга, 2
Google ученый
Крейн Д., Паскарелло Э., Джеймс Д.: AJAX в действии. 2005, Гринвич: Manning Publications
Google ученый
Скачать ссылки
Благодарности
Мы благодарим Дженнифер Р. Смит за помощь в подготовке рисунков и команду RGD. Эта работа была поддержана Национальным институтом сердца, легких и крови от имени Национальных институтов здравоохранения [HL64541].
Информация об авторе
Авторы и организации
База данных генома крысы, Центр молекулярной генетики человека и молекулярной генетики, Медицинский колледж Висконсина, Милуоки, Висконсин, 53226, США Weisong Liu, Jeff De Pons, Melinda R Dwinell & Mary Shimoyama
Авторы
- G Thomas Hayman
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Академия
- Pushkala Jayaraman
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
- Виктория Петри
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Marek Tutaj
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Weisong Liu
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
- Jeff De Pons
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Melinda R Dwinell
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Мэри Симояма
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Автор, ответственный за корреспонденцию
Г Томас Хейман.
Дополнительная информация
Конкурирующие интересы
Авторы заявляют об отсутствии конкурирующих интересов.
Вклад авторов
GTH написала рукопись и оценила программный инструмент и страницы отчета. PJ разработал приложение, изменил дизайн страниц отчета и помог написать рукопись. В.П. оценил страницы приложения и отчета и помог в написании рукописи. MT, WL и JDP участвовали в разработке программного обеспечения и изменении дизайна страницы отчета. MRD является со-главным исследователем. MS является главным исследователем и участвовал в разработке и координации заявки. Все авторы прочитали и одобрили окончательный вариант рукописи.
Оригинальные файлы изображений, представленные авторами
Ниже приведены ссылки на оригинальные файлы изображений, представленные авторами.
Оригинальный файл авторов для рисунка 1
Оригинальный файл авторов для рисунка 2
Оригинальный файл авторов для рисунка 3
Оригинальный файл авторов
67
Эта статья опубликована по лицензии BioMed Central Ltd.




 Протоколы Curr Bioinf. в прессе
Протоколы Curr Bioinf. в прессе